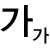
크리스퍼(CRISPR) 유전자가위 기술은 특정 유전자를 정확히 골라 높은 정밀도로 편집할 수 있는 생명공학 분야 핵심기술이다.
그러나 이 기술은 단일 유전자 정밀 편집에는 매우 효과적이지만, 다중 유전자 편집은 sgRNA(single-guide RNA) 반복 배열로 인한 기술적 제약이 따른다.
sgRNA는 카스(Cas) 단백질이 해당 유전자들에게 도달하도록 가이드 역할을 한다. sgRNA는 서열이 매우 유사하기 때문에 반복적인 sgRNA 배열은 DNA 합성의 실패율을 높이고, 플라스미드 재조합 및 발현의 불안정성을 유발해 다중 편집의 확장성과 실용화에 한계를 초래한다.
고효율 다중 유전자 편집기술
한국생명공학연구원(이하 생명연) 합성생물학연구센터 이대희 박사팀은 합성생물학 기술을 바탕으로 새로운 sgRNA 배열 방식을 개발, 유전자 편집의 효율성과 안정성을 향상시키고 기존 병목현상을 해결하는 ‘고효율 다중 유전자 편집기술’을 개발했다.
연구팀은 외부 유도물질 없이도 스스로 RNA를 절단할 수 있고, 주변 유전자의 영향을 받지 않으면서도 정확하고 안정적으로 sgRNA를 발현할 수 있는 리보자임(RiboJ) 기술을 활용했다.
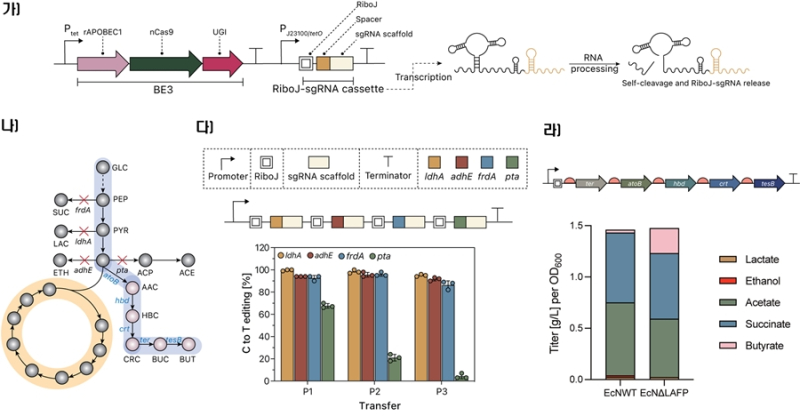
이를 통해 여러 sgRNA를 효율적으로 처리하는 ‘RAMBE(RiboJ-Aided Multiplexed Base Editing)’ 시스템과, 반복되는 유전자를 서열 없이 배열할 수 있는 ‘NR(Non-Repetitive)-RAMBE’ 시스템을 개발했다.
이 기술을 대장균에 적용한 결과 한 번에 6개 이상의 유전자를 동시에 편집해 부티르산 생산량을 최대 7배 증가시켰고, 아세트산 소비도 조절해 전체 대사경로 최적화에도 성공했다.
부티르산은 장내 미생물이 섬유질을 분해하면서 생성하는 대사산물로, 장세포의 에너지원으로 사용되고 항염증 효과와 장 건강 개선에 기여해 기능성 미생물 대사산물로 주목받고 있다.
이번 연구에 사용된 균주는 사람의 장 내 환경과 유사한 조건에서도 잘 자라는 프로바이오틱스 활용 대장균(E. coli Nissle 1917)으로, 산소가 매우 적은 환경에서도 안정적인 생산성과 편집효율을 보여 실용화 가능성을 높였다.
특히 NR-RAMBE 시스템은 기존 RAMBE보다 더욱 진화된 방식으로, sgRNA의 반복 서열이 제거돼 DNA 합성과 조립 과정이 훨씬 단순해졌고, 유전자 합성의 복잡성이 1/7로 줄어 유전자 편집 실패율도 대폭 감소했다.
이번 실험은 유전자를 동시에 6개까지 편집했음에도 기존 시스템과 비슷한 수준의 높은 편집 효율을 보여 더욱 주목받는다.
이번에 개발한 NR-RAMBE 기술은 sgRNA를 코딩하는 DNA를 안정적으로 합성할 수 있어 유전자 설계부터 합성, 검증까지 자동화하는 시스템 ‘바이오 파운드리’에 저굥에 적합해 다양한 의료·산업용 미생물 개발에도 폭넓게 활용될 것으로 기대된다.
이대희 박사는 “이번 연구는 크리스퍼 유전자가위로 여러 개 유전자를 동시에 편집할 때 가장 문제됐던 sgRNA 합성과 배열 문제를 합성생물학 기술로 해결한 것이 큰 의미를 갖는다”며 “EcN과 같은 프로바이오틱스를 포함한 치료용·산업용 미생물의 정밀 개량에 활용할 수 있고, 고효율 바이오생산 플랫폼 기술 확장도 기대된다”고 설명했다.
공동 연구책임자 이승구 박사는 “이 기술은 유전자 편집을 세포 내에서 논리적으로 조절할 수 있는 가능성을 보여준 사례”라며 “마이크로에어로빅 등 장내 환경과 유사한 조건에서도 유전적 안정성과 생산성을 유지함으로써 실제 응용 가능성을 실험적으로 검증했다”고 덧붙였다.
한편, 이번 연구결과는 지난 7일 국제학술지 ‘Chemical Engineering Journal(IF 13.4, JCR 상위 2.8%)’ 온라인에 게재됐다.
(논문명 : RiboJ-assisted non-repeated sgRNA arrays for enhanced CRISPR multiplex genome engineering in Escherichia coli / 교신저자 : 이대희·이승구 박사 / 제1저자 : 우승균·김성근 박사)